Praktikum am Max-Planck-Institut für Immunbiologie und Epigenetik in Freiburg
vom 19. bis zum 30. Oktober 2015
Von Jana Bilanovic und Alexandra Löbker
Wir, Alexandra Löbker und Jana Bilanovic aus dem 12. Jahrgang des Gymnasiums Ulricianum, durften vom 19.10.15 bis 30.10.15 ein Praktikum am Max-Planck-Institut für Immunbiologie und Epigenetik im Labor von Prof. Dr. Thomas Jenuwein in Freiburg absolvieren.
Den ersten Tag nutzten wir, um uns mit den Aufgaben der kommenden zwei Wochen vertraut zu machen und Fragen zu stellen bzw. Unklares und Unbekanntes nachzuschlagen. Außerdem mussten wir uns anfangs erst einmal gewöhnen, durchgehend Englisch zu sprechen und Inhalte auf dieser Sprache zu lesen, da im Institut Forscher aus aller Welt zusammenkommen und English dort die vorherrschende Sprache ist.
Unser erstes richtiges Projekt begann am zweiten Tag und war die Chromatin-Immunpräzipitation (ChIP). Für die Vorbereitung lösten wir erst einmal die Zellen von einer Petrischale, in welcher die Zellen gewachsen waren und sich am Boden abgesetzt hatten, mit einem bestimmten Enzym (Trypsin), wuschen sie und fixierten sie mit Formaldehyd. Dies sollte bewirken, dass alle Vorgänge quasi eingefroren werden.
Erklärend soll man sich die DNA vorstellen, die jeweils in bestimmten Abschnitten um sogenannte Histone gewickelt ist. Die Histone sind große Proteine, die jeweils Schwänzchen haben, die mit Bezeichnungen gekennzeichnet sind. Auf diesen Schwänzchen gibt es viele Positionen, an denen sich chemische Gruppen anlagern können. Diese tragen dann verschiedene Funktionen bei.
Die Stelle, die wir untersuchten, war H3K9me3. Dies bedeutet das Schwänzchen H3 an der Position K9 mit einer dreifachen Methylierung. Ist dieses H3K9me3 gegeben, wird das Gen, an welchem es angelagert ist, ein inaktives Gen. Wir wollten dann mithilfe des ChIP die DNA-Abschnitte herausfiltern, die durch H3K9me3 inaktiv sind.
Wir benutzten Wild Type (WT)-Zellen und Double Null (DN)-Zellen. Der Unterscheid zwischen den Zellen ist, dass die DN ein Enzym (Suv39h1/2) nicht besitzen. Das Enzym ist dafür da, die Reaktion zu katalysieren, die Methylgruppen an die Nukleosome bindet. Also können DN eigentlich keine (dreifache) Methylierung an der Stelle H3K9 besitzen. DN diente uns also als Negativbeispiel.
Für die weiteren Versuche bereiteten wir die Zellen vor, indem wir sie mehrfach wuschen, Stück für Stück öffneten, alles entfernten, was nicht zur DNA gehörte, und dann letztendlich nur die DNA an sich hatten. Diese wurde durch Schallwellen in kleine Stücke zerschnitten, weil die DNA so erst einmal in der endlos aufgewickelten Form vorlag und damit nicht weiter gearbeitet werden konnte.
Da wir für die ChIP eine DNA-Länge von 500 bp (Basenpaare) brauchten, mussten wir erst kontrollieren, ob die Schallwellen die DNA in richtige Stücke geschnitten hatten. Dafür nutzten wir eine sogenannte Agarosegelelektrophorese. Diese eignet sich gut, um die Länge eines DNA-Abschnitts zu bestimmen, denn die stark negativ geladene DNA wird in ein Gel geladen und an einen Stromkreis gekoppelt. Je nach Struktur ist es ihr möglich, mehr oder weniger weit durch das Gel zu „laufen“. Die Stelle an der sich die DNA nach dem Lauf befindet, lässt Aussagen über die Länge des Abschnittes zu: Denn je länger die DNA ist, desto schwerer ist der Weg durch das Gel. Die Ergebnisse haben gezeigt, dass die Abschnitte alle in der gewünschten Länge vorhanden waren (300-500bp). Danach konnte die eigentliche ChIP beginnen.
Dieses Projekt dauerte die ganze erste Woche über. Zunächst mussten die DNA-Abschnitte gefunden werden, die dieses H3K9me3 besaßen und damit auch mit dem Enzym verbunden waren, da dieses eben diese Methylierung ermöglicht.
Dafür wurde ein spezifischer Antikörper an sogenannte „magnetic beads“ gebunden. „magnetic beads“ binden in Zusammenhang mit spezifischen Antikörpern nur an bestimmte Proteine und bilden dann eine Kette. Diese Kette ist dann magnetisch und kann mit einem Magneten aus der Lösung gezogen werden. Genau das taten wir. Die Antikörper banden nur an unser Protein am H3K9me3 und so konnten wir genau die DNA-Stücke herausziehen, die unsere Bedingung erfüllten.
Hatten wir diese herausgefiltert, lösten wir die Proteine von den DNA-Abschnitten, weil wir letztendlich nur die DNA-Sequenz brauchten, um zu gucken, welches Gen inaktiviert wurde. Danach führten wir eine Real Time quantitative PCR durch. Diese Methode ist eine Darstellungsweise der Vervielfältigung bestimmter DNA-Abschnitte in Echtzeit. Da die Sequenzen der DNA-Abschnitte mit dem H3K9me3 dem Labor bereits bekannt sind, waren auch die richtigen Primer vorhanden, die für die Vervielfältigung benötigt wurden.
So konnten wir am Freitag quasi beobachten, wie sich die DNA -Abschnitte vervielfältigten und durch die Vervielfältigung war uns auch möglich zu bestimmen, wie viel von welchem DNA-Abschnitt in unserer Lösung vorhanden war.
Dazu muss noch gesagt werden, dass wir den Farbstoff „SYBR Green I“ benutzten, der als Nachweis doppelsträngiger DNA benutzt wird und Licht nur in einer bestimmten Wellenlänge absorbiert. Diese Absorption wiederum kann von einer Maschine aufgezeichnet werden, weshalb wir dann letztendlich Graphen erhielten, die uns die Menge unserer DNA-Abschnitte zeigten. Mithilfe einer Rechnung konnten wir dann tabellarisch festhalten, wie viel der durch H3K9me3 inaktivierten Gene in den Zellen vorhanden waren.
Den Anfang der zweiten Woche machte ein Western Blot. Dies ist eine typische Methode, um herauszufinden, welche Proteine sich in der Zelle befinden. Denn ähnlich wie bei der Agarosegelelektrophorese werden die Bestandteile je nach Länge oder hier dann nach Masse getrennt. Je leichter das Protein ist, desto weiter kann es im Gel laufen. Und so können die Massen der einzelnen Proteine ermittelt werden und die Masse der Proteine lässt Aussagen über die Proteine an sich zu.
Wenn dann der Lauf beendet war, konnten die Proteine auf eine Trägermembran übertragen werden. Wenn man diese dann mit zwei Antikörpern versah, wovon der zweite wieder an einen fluoreszierenden Farbstoff gekoppelt war, konnte eine spezielle Maschine die Lichtsignale empfangen, die dann indirekt von den Proteinen auf der Membran ausgingen, und sie auf eine lichtempfindliche Folie übertragen, sodass man letztendlich die Banden der Proteine deutlich auf Folie sieht.
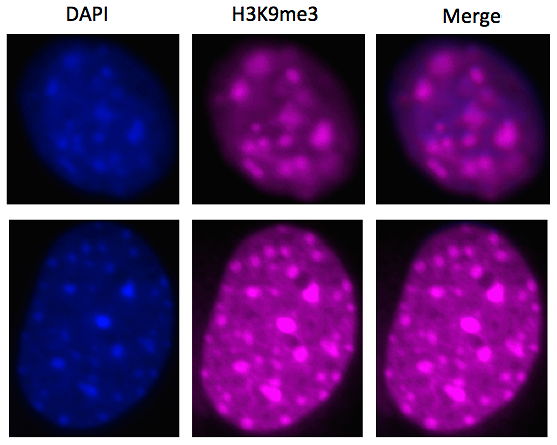
Mitte der zweiten Woche begann dann unser zweites Projekt: die Immunofluoreszenz (IF). Die Methode der IF wird genutzt, um bestimmte Zellteile für das menschliche Auge mithilfe von Farbe sichtbar zu machen. Wieder benutzten wir WT und DN Zellen, um den Unterscheid zwischen diesen Zellen deutlich aufzeigen zu können.
Für die IF würden wir zwei Farbstoffe benutzen, die uns zwei verschiedene Dinge zeigen sollten. Einmal wollten wir die Stellen aufzeigen, wo die DNA sehr eng zusammengepackt war. Diese Teile würden durch den Farbstoff DAPI gezeigt werden und blau aufleuchten. Und zum Zweiten wollten wir die Orte nachweisen, die eine Methylierung an der Stelle H3K9 aufwiesen. Für diese Stellen wurde die Farbe Cy5 genutzt, die pink leuchtet.
Da, wo die DNA sehr eng zusammen ist, findet keine Vervielfältigung der DNA statt, da dafür der offene Zustand der DNA gebraucht wird.
Für diesen geschlossenen Zustand sind H3K9me3 und H3K9me1 zuständig. Somit müssten sich bei Zellen, die das Enzym Suv39h1 besitzen die blau gefärbten Teile mit den pink Gefärbten übereinstimmen.
Zur Vorbereitung versahen wir die Zellen mit spezifischen Antikörpern, die nur an unsere Zielstrukturen banden. An sekundäre Antikörper waren unsere Farbstoffe gekoppelt. Diese banden an die primären Antikörper und wieder entstand eine Kette, die ein fluoreszierendes Ende hatte. Mithilfe eines Fluoreszenzmikroskops konnten wir dann genau die Wellenlängen des Lichts aussenden, die mit den Farbstoffen übereinstimmten.
So sahen wir unter dem speziellen Mikroskop bei beiden Zellen (WT und DN) viele blaue Stellen, wo die DNA also sehr eng zusammen war. Der Unterschied lag dann aber in den pinken Stellen, die nur an H3K9me3/1 banden. Da DN Zellen das Enzym für die Methylierung nicht besitzen, hatten sie auch keine Methylierungen an besagten Stellen, also auch keine pinke Färbung an den Stellen der geschlossenen DNA.
Diese Geschlossenheit hängt bei DN Zellen also mit anderen Stellen der Histone zusammen, die die Gene inaktivieren. Bei den WT Zellen konnte schön beobachtet werden, dass die pinken mit den blauen Stellen übereinstimmten und die Inaktivierung bestimmter Gene mit der Methylierung an der Stelle H3K9 der Histone zusammenhängt.
Alle unsere Projekte verliefen erfolgreich und die Mitarbeiter des Jenuwein-Labors standen uns stets helfend zur Seite.
Alles in allem war es ein unglaublich interessantes und lehrreiches Praktikum, das uns wissenschaftlich definitiv weiter gebracht hat. Es hat uns viele neue Bekanntschaften beschert, sowie wundervolle Austausche mit Menschen aus der ganzen Welt ermöglicht.
Wir möchten uns sehr bei den Auricher Wissenschaftstagen und den dafür zuständigen Lehrkräften sowie bei Herrn Jenuwein für die Ermöglichung dieser tollen zwei Wochen bedanken.