Praktikum am Max-Planck-Institut für Entwicklungsbiologie in Tübingen
vom 8. bis zum 12. Oktober 2018
Von Linda El Masri
Im Rahmen der Auricher Wissenschaftstage habe ich vom 08.10. bis zum 12.10.2018 an einem einwöchigen Praktikum am Max-Planck-Institut für Entwicklungsbiologie in Tübingen teilgenommen.
Der Bereich der Genetik des MPIs, in welchem ich mir einen Einblick verschaffen durfte, beschäftigt sich mit der Entwicklung und Evolution der Zebrafische (Danio rerio). Man hat sich zum Ziel gesetzt, die evolutionsabhängigen Veränderungen der Muster der Fische herauszufinden und diese zu erforschen. Im Laufe dieses Berichtes werde ich die einzelnen Forschungsmethoden beschrieben, welche das MPI für Entwicklungsbiologie verwendet. Zugleich werde ich meine Tätigkeiten genauer erläutern.
Am Montag, den 08.10.2018 wurde ich morgens von Horst Geiger empfangen und mit den Räumlichkeiten und den Mitarbeitern des Max-Planck-Ringes 5 bekannt gemacht. Nachdem dies erfolgt war, wurde ich in die Abteilung III für Genetik, welche unter der Leitung von Prof. Dr. Christiane Nüsslein-Vollhardt steht, geführt. Diese wird auch „Fischhaus“ genannt und würde mein zukünftiger Arbeitsplatz sein.
Zwischen einigen Laboren gab es auch Zimmer, in denen die Fische aufgezogen und untergebracht wurden. In Raum 1 wurden die Fische untergebracht, welche sich noch im „Babyalter“ befinden. Raum 6 wiederum bietet den älteren Fischen eine Unterkunft. Versorgt werden diese Räume durch ein spezielles Filter- und Wasseraufbereitungssystem, bei welchem Leitungswasser verschiedene Stationen durchläuft, um mit Nährstoffen angereichert zu werden und um so zu den Tanks zu gelangen. Läuft das verbrauchte Wasser ab, so gelangt es in einen Bio-Filter, in dem sich bestimmte Bakterien anreichern und das Wasser ebenfalls reinigen.
Nach dem Rundgang wurde eine Sicherheitsbelehrung durchgeführt – um mir die Gefahren und Risiken bei Laborarbeiten bewusst zu machen und um andere durch meine Arbeit nicht zu gefährden.
Direkt danach ging es auch schon in einen weiteren Teil der Fischanlage, in dem Herr Geiger und ich jeweils zwei Zebrafischpaare in einen Behälter (auch Mouse Cage genannt) zusammengesetzt haben. Zebrafische haben jedoch die unangenehme Eigenschaft, ihre eigenen Eier zu fressen. Infolgedessen wurden Fische und Eier durch ein Gitter voneinander getrennt.
Neben dem Fischprojekt starteten wir eine Polymerase-Kettenreaktion (PCR), mit welcher man herausfinden kann, welche Gene für eine bestimmte Mutation verantwortlich sind. Hierbei wurden ausgewählte Fische genommen, bei denen eine Hautprobe mit einem Wattestäbchen entnommen wurde. Diese DNA-Probe wurde gemeinsam mit Primer, Polymerase, Nukleotiden und Natronlauge in einem kleinen Behälter gemischt und in einen sogenannten „Cycler“ gegeben. In diesem Gerät wird das DNA-Gemisch erhitzt, sodass sich der DNA-Doppelstrang voneinander löst (denaturiert) und einsträngig wird. Nach und nach wird die Temperatur abgekühlt und danach wieder erhitzt, damit die Primer und Polymerase sich an die Stränge ansetzten und einen neuen DNA-Strang bilden können. Dieser Vorgang wiederholt sich so lange, bis die gewünschte Stelle in der DNA nur noch in einem kleinen Doppelstrang vorhanden ist.
Wenn dieser Vorgang erfolgt ist, geht es mit der Gelelektrophorese weiter, welche Herr Geiger und ich am nächsten Tag, dem Dienstag, weitergeführt haben. Hierzu wurde ein Agarosegel gegossen und in einen kleinen Tank mit Salzlösung gelegt. Diese Salzlösung leitet die Elektrizität weiter, welche die einzelnen Teile der DNA voneinander trennen soll. Damit die DNA-Probe auch gut zu erkennen ist, wird ein farbiger Puffer zu der Probe gegeben und in eine Geltasche, mithilfe einer Pipette, gefüllt. Die DNA ist aufgrund der elektrischen Ladung und der Größe seiner einzelnen Bausteine zu den Polen gewandert. Durch eine Fotografie des Gels kann man anschließend mit der Analyse beginnen.
Nachdem auch dieser Vorgang gelungen war, führten Herr Geiger und ich das Cycle-Sequencing durch. Beim Cycle-Sequencing werden Teile der DNA sequenziert. Dies erfolgt (wie bei der PCR) mithilfe der DNA, Primer, Nukleotide und Polymerase und einer weiteren Art von Nukleotiden, den Didesoxynukleotiden (didNTP). Das DNA-Gemisch wird nun im Cycler erhitzt, damit sich die Doppelstränge voneinander lösen. Ist dies erfolgt, so wird die Probe abgekühlt, damit sich der Primer an die DNA ansetzen kann.
Damit sich das Enzym Polymerase an den DNA-Strang ansetzen und einen neuen Strang bilden kann, welcher komplementär zum originalen ist, wird die Probe ein weiteres Mal erhitzt. Die Polymerase synthetisiert die DNA mithilfe der Nukleotide. Jedoch wird die Synthese durch ein didNTP abgebrochen – aus diesem Grund gibt es DNA-Stränge, welche unterschiedlich lang sind. Um die Ergebnisse auszuwerten, übergaben wir einigen Kolleginnen aus dem Labor das sequenzierte Gemisch.
Als die Sequenzierung getan war, schauten wir, ob die Fische, welche wir am Tag zuvor zusammengesetzt hatten, Eier gelegt haben. Gemeinsam mit Silke Geiger-Rudolph legten wir die Fischeier in einer Petrischale unter ein Binokular und trennten befruchtete und unbefruchtete Eier voneinander. Da die unbefruchteten Eier bei ihrem Faulungsprozess den befruchteten Eiern den Sauerstoff entnehmen, werden diese frühzeitig „entsorgt“.
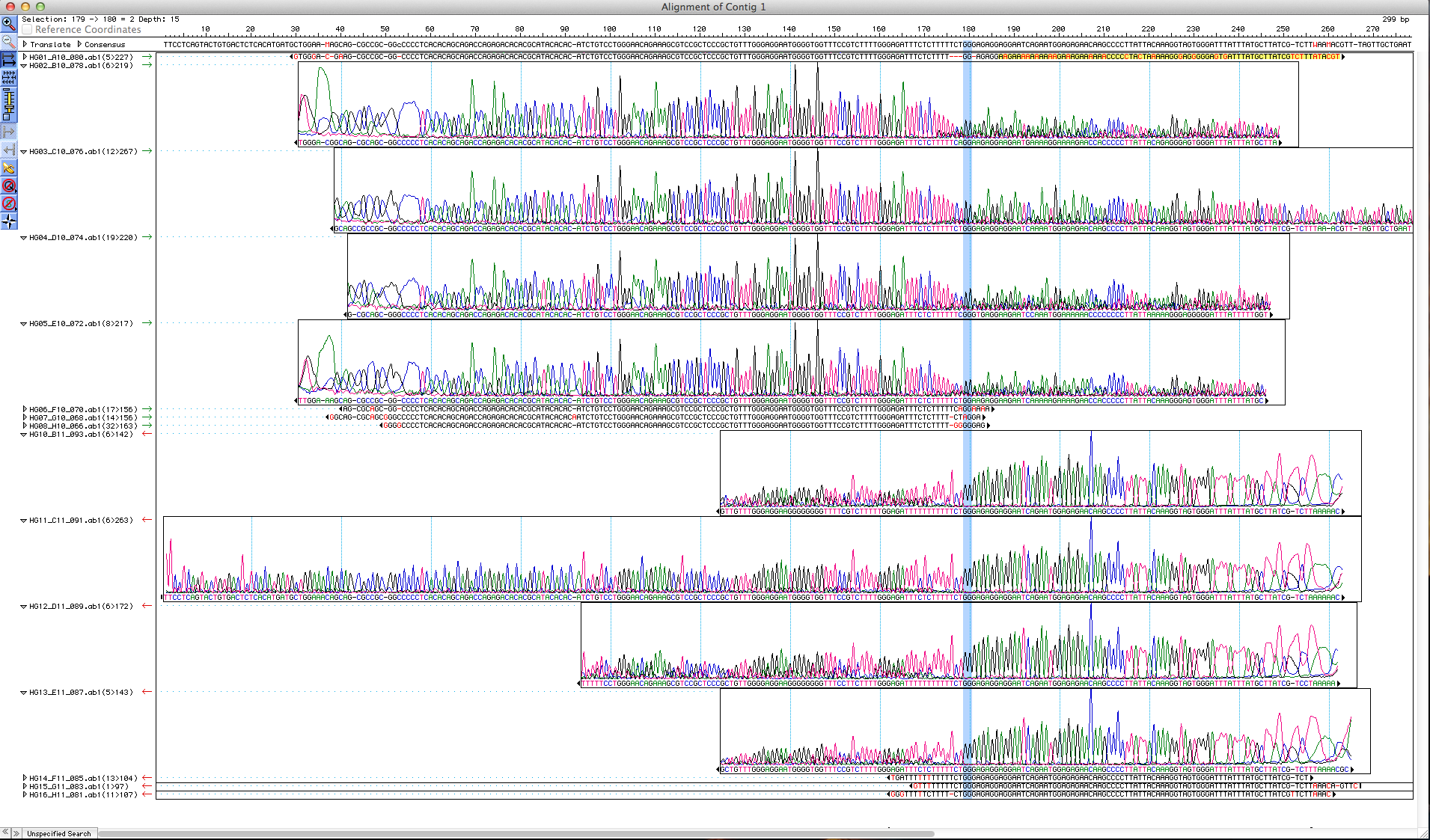
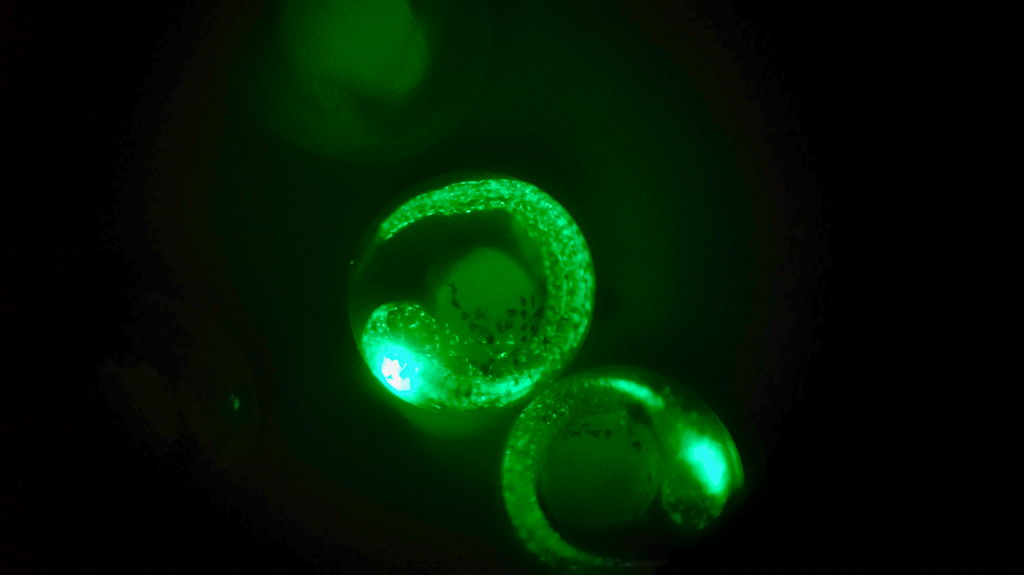
Am Mittwoch untersuchten wir die von uns angesetzten Eier unter einem Fluoreszenzmikroskop (Mikroskop mit UV-Licht), nach GFP (grün fluoreszierendes Protein) und RFP (rot fluoreszierendes Protein). Aufgrund des Lichtspektrums erfolgt die Untersuchung nach GFP mit blauem Licht, während RFP mithilfe von grünem Licht erkannt werden kann. Je nachdem, ob eine der Strukturen der Zebrafische fluoreszierend markiert ist, leuchten die Strukturen hell auf. Diese Strukturen werden von einem bestimmten Gen geschaffen. Wenn also ein Teil des Körpers markiert ist und leuchtet, dann ist es nicht das Gen selbst, sondern die Stelle, wo das Gen exprimiert wird und die Information des Gens in Erscheinung tritt.
Dieser Vorgang nahm sehr viel Zeit in Anspruch.
Am Donnerstag verbrachte ich meinen Arbeitstag gemeinsam mit Mario, dem Tierpfleger. Mario brachte mir die grundlegenden Dinge zur Haltung von Zebrafischen nahe. Die Fische werden drei Mal täglich gefüttert. So werden sie morgens und abends mit Flockenfutter und mittags mit Artemien (kleinen Krebsen) gefüttert. Zwischendurch haben wir immer wieder die Fischtanks kontrolliert – dass das Wasser vernünftig läuft oder bereits verstorbene Fische aus dem Tank gefischt wurden.
Der Freitag war für mich sowohl letzter Arbeitstag als auch Abreisetag. Die letzten zwei Stunden vor meiner Abreise verbrachte ich damit, noch einmal gemeinsam mit Herrn Geiger die Fischeilarven auf GFP und RFP zu untersuchen.
Durch das Praktikum am MPI für Entwicklungsbiologie habe ich sehr viele neue Erfahrungen sammeln können und mir wurde ein großer Einblick in die Vielfalt der Biologie und der zugehörigen Forschung gewährt.
An dieser Stelle möchte ich mich recht herzlich bei meinen Betreuern Horst und Silke Geiger bedanken, welche sich viel Zeit für mich nahmen und mir alles in Ruhe erklärt haben. Ebenfalls danke ich der BBS 2 Aurich sowie den Auricher Wissenschaftstagen, dass mir dieses Praktikum ermöglicht wurde.