Von Bosse Großmann und Jann Eden
20.07.2025 bis zum 01.08.2025


Nachdem wir am Sonntag, dem 20.07.2025 mit dem Zug in Heidelberg angekommen waren und uns in der Jugendherberge eingefunden haben, konnten wir am darauffolgenden Montag mit dem Praktikum im Life-Science Lab beginnen.
Am Montag erwartete uns unsere Praktikumsleiterin Anja Klefenz und ihre Kollegin Angela Funk. Nach einer kurzen Begrüßung und der daran anschließenden Vorstellungsrunde erhielten wir eine Sicherheitseinweisung. Im Fokus dabei lag unser S1-Labor, in welchem wir die nächsten beiden Wochen verbringen sollten. Dort wurden wir mit den grundlegenden Arbeitstechniken und Geräten vertraut gemacht. Neu für uns waren die Eppendorfpipetten, die eine hohe Präzision gewährleisten.

Vom Mittagessen der hauseigenen Kantine gestärkt begannen wir mit den Vorbereitungen für die diskontinuierliche Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese (SDS PAGE), einem Verfahren, das das Auftrennen von Proteinen entsprechend der Größe ermöglicht. Dazu stellten wir zunächst ein Trenn- und ein Sammelgel her, welche wir in einem Gießstand schichteten. Das Sammelgel erfüllt dabei die Funktion, die zu untersuchenden Proteine zu konzentrieren, um sie daraufhin besonders gut im Trenngel aufspalten zu können. Hierin sollten dann später die verschiedenen Banden der Proteine sichtbar werden. Um den Einfluss der Faltstruktur und der Ladung der Proteine zu verhindern, wurde zum Gel jeweils SDS gegeben, welches die Eigenladung überdeckt und die Proteine entfaltet. Das Ziel hiervon war es, im späteren Verlauf des Praktikums ein Auftrennen ausschließlich nach Molekülgröße zu ermöglichen.
Anschließend stellten wir Agar her, welcher als Nährmedium für die Bakterien während der Untersuchungen der Bakterienkulturen in den nächsten Tagen dienen sollte.
Am Dienstag haben wir mit den SDS-Gelen des Vortags weitergearbeitet. Dazu stellten wir zunächst verschiedene Proben her, die ein zu untersuchendes Proteingemisch enthielten. Darunter waren vier Proben, welche jeweils mit verdünnten BSA befüllt wurden. Die Konzentration des BSA war uns bekannt. Außerdem fertigten wir eine fünfte Probe an, die ein Proteingemisch mit unbekannter Konzentration, das sogenannte FCS, enthielt. Nach dem Zentrifugieren und Erhitzen erhielten wir Proteine, die aufgrund dieser Schritte nur noch in ihrer Primärstruktur vorlagen. Dadurch lassen sich die Proteine der Größe nach auftrennen. Dann pipettierten wir diese fünf Proben und einen zusätzlichen Marker in die Kammern des Vertikalgels. Über die Mittagspause legten wir eine Spannung an, wodurch die Proben durch das Gel wanderten. Die Wartezeit überbrückten wir des Weiteren mit dem Gießen von Agarplatten aus den schon vorbereiteten autoklavierten Flaschen, welche den Agar beinhalteten. Bevor wir den Agar auf die Petrischalen gossen, fügten wir das Antibiotikum Kanamycyn der Lösung hinzu. Außerdem stellten wir Platten her, die das Antibiotikum Ampicillin enthielten. Die Petrischalen wurden danach in den Kühlschrank gestellt, damit der darin enthaltene Agar aushärten konnte.
Nach dem Mittagessen setzen wir die Arbeit an unseren SDS-Gelen fort, welche wir nun mithilfe eines Spatels lösten und das Sammelgel vom Trenngel separierten. Das Trenngel legten wir in eine kleine Plastikschale und gaben zum Anfärben Comassie-Färbelösung hinzu. Die Schale deckten wir luftdicht mit Parafilm ab und stellten sie auf einen Schüttler. Dann widmeten wir uns einem weiterten Verfahren, der Proteinbestimmung nach Bradford. Dieses Verfahren ermöglicht eine Konzentrationsbestimmung einer unbekannten Proteinlösung, in unserem Fall der FCS-Probe. Bei dem Verfahren macht man sich die Absorptionsänderung eines Farbstoffs, hier Roti-Quant-Färbelösung, in Abhängigkeit zur Proteinmenge zu Nutze. Zunächst erstellten wir hierzu eine Standardreihe beziehungsweise eine Kalibrierungsgerade mit acht Proben, die eine unterschiedliche Konzentration an BSA enthielten. Die Proben untersuchten wir dabei mit einem Spektralphotometer und übertrugen die Werte in einen Graphen. Dadurch war es uns möglich, eine lineare Regression durch die Werte zu legen. Auch die unbekannte Probe mit FCS ließen wir vom Spektralphotometer untersuchen und konnten durch die Kalibrierungsgerade des BSA nun seine Konzentration ablesen.
Den Abschluss des Tages bildete das Gießen von Agarosegelen für eine spätere Agarosegelelektrophorese mithilfe eines Horizontal-Gels. Diesen Schritt hat Frau Klefenz für uns übernommen.
Der Mittwoch begann für uns mit dem Entfärben der Proteingele, wodurch die Banden der einzelnen Proben sichtbar wurden, sodass wir diese fotografieren konnten.
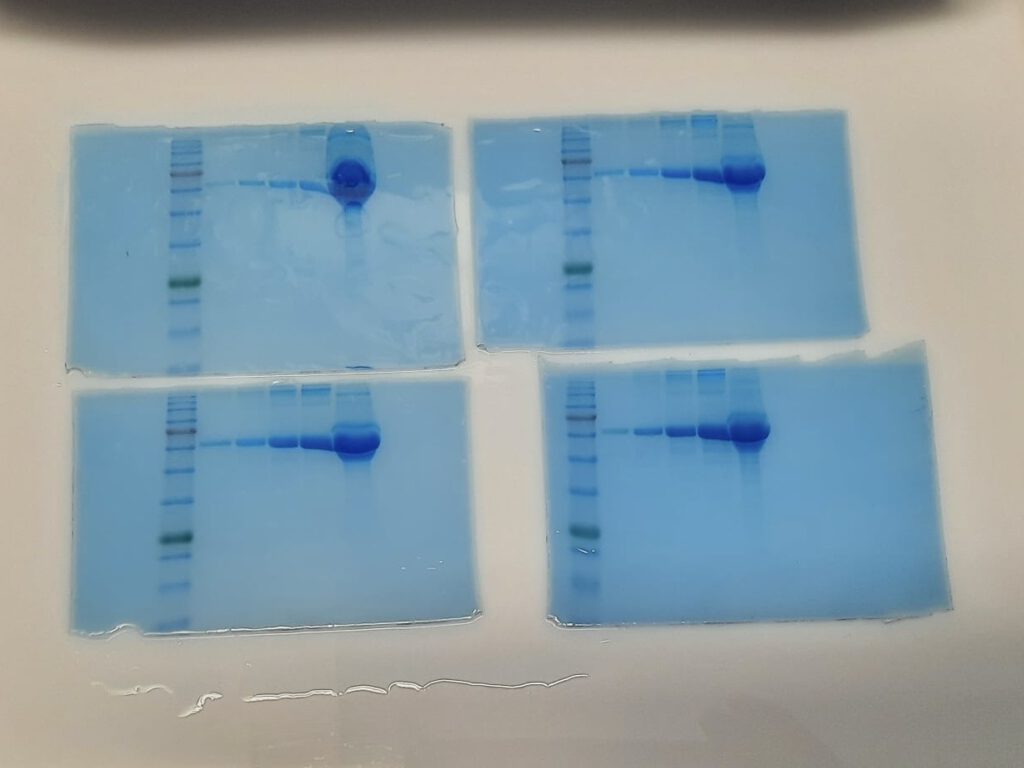
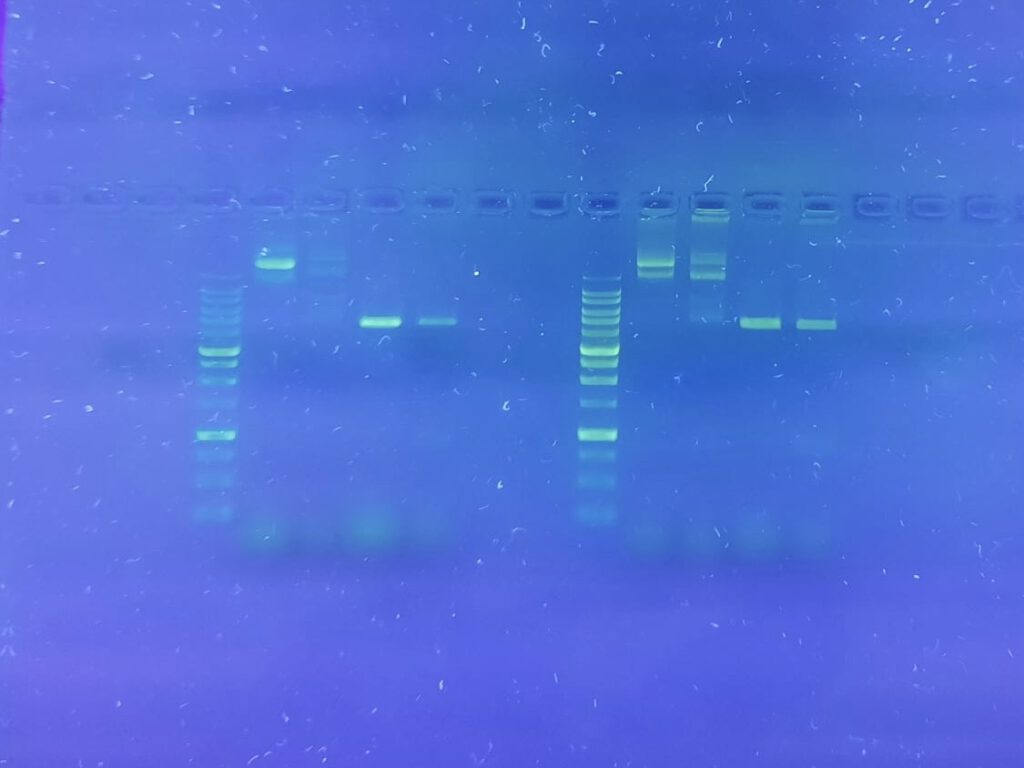
Des Weiteren beschäftigten wir uns am Vormittag mit dem Anfertigen einer sogenannten Minipräp, einer Methode der DNA-Isolierung im kleinen Maßstab. Dazu wurden bereits am Vortag Bakterienkulturen angeimpft. Uns wurden zwei verschiedene Bakterienkulturen gegeben, die entweder das Plasmid pEGFP-N1 oder das Plasmid pEGFP-N1 JAM enthielten. Das zweite Plasmid enthielt hierbei eine Zusatzsequenz von ca. 900 Basenpaaren. Mithilfe verschiedener Pufferlösungen und mehrfachem Zentrifugieren isolierten wir die Plasmid-DNA von den restlichen Zellbestandteilen. Durch Fällen, Waschen und Trocknen konzentrierten und entsalzten wir die Plasmid-DNA. Diese lösten wir daraufhin in destilliertem Wasser. Nach der Mittagspause setzten wir die Arbeit mit unserer Plasmid-DNA fort, indem wir einerseits eine Transformation und andererseits eine Restriktionsanalyse durchführten. Für die Restriktionsanalyse nutzten wir die Restriktionsenzyme HindIII und BamHI, sowie einen Puffer, destilliertes Wasser und jeweils eine unserer zwei Plasmid-Lösungen. Aus diesen fertigten wir zwei restriktionsverdaute Ansätze an. Diese verglichen wir mit den „ungeschnittenen“ Proben, indem wir sie gemeinsam mit einem Marker durch das am Vortag vorbereitete Agarosegel laufen ließen. Indem wir zu den Proben jeweils DNA-Ladepuffer hinzugaben, wurden die Ergebnisse unter UV-Licht sichtbar. Die ungeschnittenen Plasmide lieferten dabei keine klaren Banden, die geschnittenen Ansätze zeigten eine bzw. zwei klare Banden.
Die Methode der Transformation ermöglichte außerdem, die isolierten Plasmide in chemisch kompetente, also besonders aufnahmefähige Bakterien einzubringen. Nach Zugabe unserer Plasmide gemeinsam mit einem Puffer und destilliertem Wasser zu den kompetenten Bakterien, ließen wir diese mit zusätzlicher Nährflüssigkeit im Wärmeblock inkubieren. Abschließend gaben wir unsere jeweiligen Proben mit den verschiedenen Plasmiden auf die im Laufe der Woche vorbereiteten Agarplatten, welche entweder das Antibiotikum Kanamycin oder Ampicillin enthielten. Außerdem gaben wir eine verdünnte Probe auf eine Kanamycin-Platte. Um die Vermehrung der Bakterien auf den Agarplatten zu garantieren, stellten wir diese über Nacht in den Inkubator, der die optimalen Bedingungen gewährleistet. Am Donnerstag widmeten wir uns der Zellbiologie. Begonnen haben wir mit dem Kultivieren der HeLa-Zellen (Gebärmutterhalskrebszellen). Hierbei war das sterile Arbeiten besonders wichtig, um die Kontamination der Bakterienkultur zu vermeiden. Dazu arbeiteten wir an speziell dafür vorgesehenen Sterilwerkbänken. Nach gründlichem Waschen und Zentrifugieren der Zellkultur überführten wir diese nach einem Splitfaktor in eine neue Zellkulturflasche und resuspendierten sie in einem neuen Medium. Unsere neu kultivierten HeLa-Zellen lagerten wir über das Wochenende im Inkubator.

Am Nachmittag führten wir eine neue Methode durch, die Ermittlung der Zellzahl mit der Neubauer-Zählkammer. Hierzu pipettierten wir Jurkat-Zellen (Leukämie-Zellen) unter das Deckgläschen der Zählkammer und zählten die Zellen unter dem Mikroskop, um deren Konzentration in der Lösung zu bestimmen.

Bei den Jurkat-Zellen induzierten wir daraufhin die Apoptose durch Zugabe eines Todesliganden und die Nekrose durch Erhitzen der Probe auf dem Wärmeblock. Die Proben gaben wir auf eine 96-Well-Platte. Mithilfe unterschiedlicher Mengen des dazugegebenen Todesliganden wollten wir am nächsten Tag die Prozesse der Apoptose und der Nekrose genauer untersuchen. Am Schluss des Tages führten wir die Transfektion von Hek 293T Zellen (human embrionic kidney cells) durch, indem wir die Calciumphosphat-Methode, ein Verfahren zur Einschleusung von DNA in eukaryotische Zellen, anwandten. Das Gemisch aus Plasmid pGFP-N1, Calciumchlorid und einem Puffer gaben wir nach dem Invertieren, Zentrifugieren und Inkubieren tropfenweise zu den HekZellen.
Außerdem sahen wir uns die Bakterienkulturen auf den Agarplatten an. Dabei war festzustellen, dass auf allen Platten Bakterienkulturen wachsen konnten. Eigentlich sollte dies nicht der Fall sein, da die Bakterien nicht über ein Ampicillinresistenz-Gen verfügen. Jedoch ist es möglich, dass das Antibiotikum bei zu hoher Temperatur zum Agar geben wurde, wodurch es denaturiert und nicht mehr funktionsfähig war.
Zum Abschluss der Woche untersuchten wir die Proben der Jurkat-Zellen auf der 96-Well-Platte mit einem Mikroskop und einem Durchflusszytometer. Beim Mikroskopieren achteten wir vor allem auf morphologische Unterschiede der Zellen, um zu identifizieren, wie groß der Anteil der lebenden Zellen war. In Abhängigkeit von der Menge des Todesliganden, die wir zu den Proben gegeben hatten, waren mehr oder weniger lebende Zellen vorhanden. Auch die Probe ohne Todesligand wies apoptotische Zellen auf, da die Apoptose als natürlicher Prozess weiterhin abläuft. Die Apoptose wird dadurch sichtbar, dass das Zellvolumen schrumpft und in apoptotische Körper zerfällt. Die Probe, die auf 70°C erhitzt worden war (Nekrose), unterschied sich insofern von allen anderen Proben, als dort keine lebenden Zellen übrig waren, da die Zellen durch die Temperatur „aufgeplatzt“ waren. Diese Beobachtungen verifizierten wir mit dem Durchflusszytometer, welches mithilfe eines Laserstrahls erkennen kann, wie groß der Anteil lebender Zellen ist.

Zusätzlich sahen wir uns unsere Hek-Zellen an, die durch die Transfektion das Plasmid pGFP-N1 aufgenommen haben sollten. Um zu überprüfen, ob die Zellen die Plasmide erfolgreich aufgenommen hatten und das grün fluoreszierende Protein exprimierten, betrachteten wir diese unter einem Fluoreszenzmikroskop. Dort wurden grüne Zellen sichtbar, was bedeutete, dass wir unsere Plasmide erfolgreich in die Hek-Zellen eingebracht hatten.
Erholt starteten wir am Montag im DKFZ-Haupthaus in die neue Woche. Die zweite Woche war so aufgebaut, dass wir am Vormittag Besuche in den verschiedenen Abteilungen vornahmen und am Nachmittag unsere Arbeit im Labor fortsetzten. Der erste Besuch führte uns ins Zentrum für präklinische Forschung. Vor Ort wurden wir freundlich von Frau Dr. Lena Hornetz begrüßt, die uns das Tierhaus vorstellte. Dort befinden sich ca. 35.000 Mäuse, die von den rund 55 Tierpflegern sorgsam und gewissenhaft gepflegt werden. Die Mäuse dienen hierbei in erster Linie der Erforschung von Krebs und verschiedenen Tumoren. Durch den Besuch wurde uns deutlich, dass der oftmals negativ behaftete Begriff „Tierversuch“ nicht auf die heutige Arbeit mit Tieren in der Forschung übertragbar ist. Denn um mit Tieren forschen zu können, werden strikte Richtlinien befolgt, Kontrollen durchgeführt und Genehmigungen angefordert, um das Wohlergehen der Tiere zu gewährleisten.
Anschließend begrüßte uns Armin Runz in der Abteilung Medizintechnik und führte uns in die grundlegenden Ziele und Methoden ein. Im Fokus standen dabei die 3D-Drucker, deren Ziel es ist, den menschlichen Körper und dessen Organe nachzubilden, um sie besser zu verstehen. Bevor ein menschlicher Tumor bestrahlt wird, ist es durch die Medizintechnik möglich, diesen zu modellieren und die Strahlenbelastung zu messen. Hierfür ist es wichtig, dass die 3D-Drucker mit Materialien verschiedener Dichte arbeiten, um Gewebe im CT und MRT darstellen zu können. Abschließend präsentierte Herr Runz uns die Geräte sowie einige gedruckte Modelle. Er freute sich insbesondere, uns seinen neuesten 3D-Drucker zeigen zu können, der eine noch präzisere und umfangreichere Medizintechnik ermöglichen soll.

Zurück im Labor widmeten wir uns nachmittags der Arbeit mit unseren HeLa-Zellen an den Sterilwerkbänken. Die zuvor umkultivierten HeLa-Zellen plattierten auf einer 6-Well-Platte aus und stellten sie in den Inkubator.
Am nächsten Tag stellte uns Dipl.-Physiker Jens Lang die Stabsstelle Strahlenschutz und Dosimetrie vor. Er erklärte uns die grundlegenden Arten von Strahlungen und deren Anwendung am DKFZ. Die verschiedenen Strahlungen finden dabei sowohl in der Krebstherapie am Patienten als auch in der Forschung in den Laboren Verwendung. Hierbei ist besonders auf die Halbwertszeit der Stoffe zu achten, um eine übermäßige Strahlenbelastung der Patienten, der Mitarbeiter und der Umgebung zu vermeiden.
Der nächste Besuch führte uns in die Abteilung der Durchflusszytometrie, in der Dr. Marcus Eich die Funktionsweise der Durchflusszytometer erklärte. Diese dienen neben der Identifikation und Vermessung von Zellen ebenfalls dem Sortieren verschiedener Zellarten. Hierzu binden Antikörper, die mit Fluoreszenzfarbstoffen markiert werden, an zellspezifische Oberflächenproteine. Die Zellen werden daraufhin im Durchflusszytometer von einem Laser bestrahlt. Dadurch entsteht ein Streulicht, welches es ermöglicht, den Zelltyp zu identifizieren. Zusätzlich kann anhand der Wellenlänge auf den Fluoreszenzfarbstoff und somit auf die Art der Zelle geschlossen werden. Anschließend können die Zellen durch ein elektrisches Feld für die weitere Verwendung sortiert werden.

Als wir nachmittags wieder im Life-Science-Lab angekommen waren, holten wir die HeLa-Zellen aus dem Inkubator und gaben nach mehrfachem Waschen Färbelösungen hinzu. Bei der einen Hälfte der Proben sollten nur die Zellkerne durch DAPI-Färbelösung angefärbt werden, wohingegen bei der anderen Hälfte auch die Mitochondrien durch Mito-Tracker-Red angefärbt werden sollten. Abschließend gaben wir die ausplattierten und angefärbten Zellen auf einen Objektträger und ließen diesen abgedeckt über Nacht trocknen.
Am nächsten Morgen waren wir beim Krebsinformationsdienst zu Besuch. Dort hieß uns zunächst Frau Dr. Juliane Folkerts willkommen, die uns über Krebs bei Kindern und Jugendlichen aufklärte. Zu den häufigsten Tumorarten in dieser Altersgruppe zählt Leukämie, bei der das blutbildende System betroffen ist. Durch verschiedene Behandlungsmethoden wie Chemotherapie, Operation oder Immuntherapie sind die Heilungschancen für Krebs bei Kindern oftmals hoch. Dennoch wird an neuen Methoden geforscht, dem Krebs entgegenzuwirken und die Patienten dabei möglichst wenig zu belasten. Hierzu stellte uns Frau Folkerts die Methode der bispezifischen Antikörper und die CAR-T-Methode vor. Bei dieser werden körpereigene T-Zellen derartig modifiziert und dann vervielfältigt, dass sie die betroffene Zellart erkennen und gezielt zerstören können. Danach besuchten wir Frau Dr. Rotraut Fischer, die Teil des Telefonteams des Krebsinformationsdienstes ist. Die Mitarbeiterinnen und Mitarbeiter haben rund 362 Tage im Jahr von 08:00 Uhr bis 20:00 Uhr ein offenes Ohr für die Menschen und vermitteln ihnen sachliche und evidenzbasierte Informationen rund um das Thema Krebs. Durch eine riesige Datenbank ist es ihnen möglich, den jährlich rund 24.000 Anrufern neutrale und individuelle Auskünfte zu geben. Der Krebsinformationsdienst steht allen bei Fragen, Sorgen und Unklarheiten sowohl per Email als auch unter der Telefonnummer 0800 420 3040 zur Verfügung. Beim Besuch wurde uns bewusst, wie wichtig diese Arbeit für die Gesellschaft ist. Vor dem Mittagessen besuchten wir außerdem die Aquahaltung, in der rund 400 südafrikanische Krallenfrösche leben. Tierpflegerin Melina Fichtmüller zeigte uns einige Frösche und erklärte, dass nur der Froschlaich, nicht aber die Frösche selber zur Forschung verwendet werden. Am Nachmittag begannen wir unser letztes Laborprojekt, die PCR (Polymerase-Kettenreaktion). Für die semi-quantitative PCR setzten wir eine Verdünnungsreihe mit GFP-Plasmiden an und pipettierten Primer, Wasser, unsere verschiedenen Verdünnungsansätze und ein Gemisch aus dNTP, Puffern und Taq-Polymerase in die PCR-Tubes. Abschließend stellten wir sie in einen Thermocycler, welcher die verschiedenen Schritte Denaturierung, Annealing und Elongation in mehreren Zyklen durchführte. Die andere Art der PCR, die wir durchführten, war die Colony-PCR, bei welcher wir statt isolierter DNA Bakterienzellen verwendeten. Auch diesen Ansatz stellten wir in einen PCR-Block.
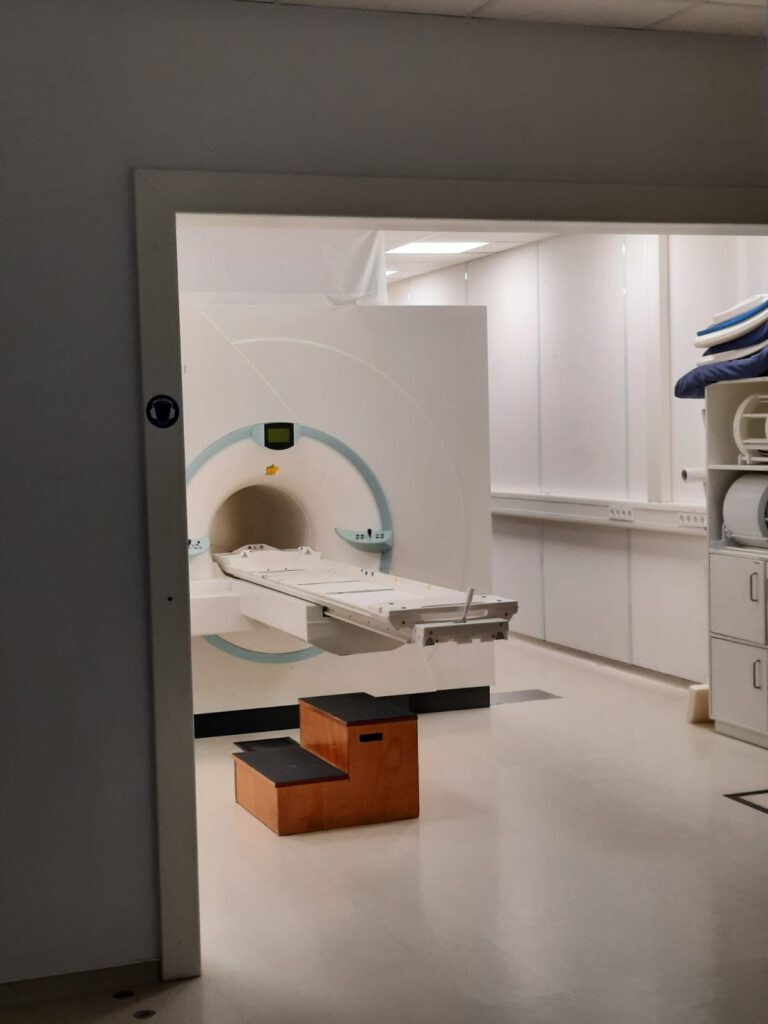
Unsere letzten Abteilungsbesuche fanden am Donnerstag statt. Am Vormittag besichtigten wir den 7-Tesla-Resonator, ein Gerät, welches Magnetresonanztomographien (MRT) und Spektroskopien durchführen kann. Dr. Stephan Orzada erläuterte uns die Grundlagen eines MRTGeräts und verglich dabei die verschiedenen Feldstärken 1,5 bzw. 3 und 7-Tesla. Ein 7-Tesla Gerät bietet den Vorteil, dass eine höhere Auflösung des zu untersuchenden Gewebes möglich ist. Im Anschluss durften wir das Gerät besichtigen und mithilfe einer Aluminium-Platte, eines Tennisballs, einem Stück Alufolie und eines Stromkabels selber herausfinden, welchen Einfluss das Magnetfeld auf die Umgebung hat.
Die letzte Abteilung, die wir besuchten, war die Hochdurchsatzsequenzierung, in welcher uns Dr. Angela Schulz erläuterte, welche Vorteile diese Methode bietet und wie sie im Detail abläuft. Durch diese Methode kann das menschliche Genom basierend auf PCR innerhalb von 2 Tagen ausgelesen werden. Dies macht man sich insbesondere bei der Krebsforschung zunutze, da dadurch untersucht werden kann, welche Veränderung im Genom zur Entstehung des Tumors geführt hat. Hierzu wird die DNA „gesunder“ Zellen des Patienten mit der DNA von Tumorzellen verglichen. Indem hierdurch die genetische Ursache der Krebsentstehung sichtbar wird, lässt sich eine wesentlich personalisiertere Therapie durchführen. Somit spielt die Abteilung Hochdurchsatzsequenzierung eine Schlüsselrolle in der modernen Krebsforschung. Um den Prozess der Sequenzierung besser verstehen zu können, verfolgten wir im Anschluss den Weg der Proben im Labor und hatten somit die Möglichkeit, die Geräte und insbesondere die Hochdurchsatzsequenzierer zu betrachten.

Am letzten Tag werteten wir unsere letzten Experimente im Labor aus. Wir bereiteten die PCRProben für eine weitere Gelelektrophorese in einem Agarosegel vor, indem wir einen Farbstoff hinzugaben. Während wir die Proben unter Spannung im Gel laufen ließen, begutachteten wir unsere auf Objektträgern fixierten Zellen unter einem Fluoreszenzmikroskop. Die Funktionsweise eines Fluoreszenzmikroskops wurde uns von Dr. Norbert Mücke näher gebracht. Bei der Betrachtung unter dem Mikroskop wurden durch die Fluoreszenzfarbstoffe DAPI und Mito-Tracker die Zellkerne und Mitochondrien sichtbar.
Zuletzt schauten wir uns unsere Agarosegele an, bei welchen nun deutlich wurde, dass die PCR bei allen Proben ordnungsgemäß ablief, da die Proben im Vergleich zur nicht vervielfachten DNA eindeutige Banden aufwiesen. Nach einem letzten Mittagessen in der Kantine des DKFZ verabschiedeten wir uns und traten die Heimreise nach Aurich an.
Abschließend möchten wir noch die Gelegenheit nutzen, uns bei den Auricher Wissenschaftstagen für die einmalige Möglichkeit des Praktikums im Life-Science Lab des DKFZ in Heidelberg zu bedanken. Wir freuen uns, dass wir diese Erlebnisse machen durften. Nicht zu vergessen sind natürlich Anja Klefenz und Angela Funk, die uns während des gesamten Praktikums begleitet haben. Ihre umfassende Vorbereitung und ihr bemerkenswertes Engagement machten das Praktikum zu einer einzigartigen Erfahrung für uns. Des Weiteren bedanken wir uns herzlich bei den Abteilungen des DKFZ für die spannenden und informativen Einblicke in ihre Arbeit, insbesondere Dr. Lena Hornetz, Armin Runz, Dipl.-Phys. Jens Lang, Dr. Marcus Eich, Dr. Juliane Folkerts, Dr. Rotraut Fischer, Melina Fichtmüller, Dr. Stephan Orzada, Dr. Angela Schulz und Dr. Norbert Mücke.










